申請書作成に当たっての注意点
これまでに審査員から指摘される頻度が多かった内容です。
・NGSにより目的が達成できるのかどうかの検討が不十分
( 例えば、RNA-seqの比較だけで注目する形質の原因遺伝子を見つける、というような申請は
説明不足になりがちです。 )
・サンプル準備に対する計画性や妥当性
・非モデル生物等のリファレンスゲノムが適用されにくい場合の解析方針
( 例えば、変異解析を行う場合にはデータをどのように扱うのか、解釈するのかを十分に
説明する必要があります。 )
解析規模の目安
当センターが提案している1サンプル当たりの解析規模です。
| 解析規模の目安 | |
| RNA-seq ( モデル生物 ) | 5M~20Mリード |
| RNA-seq ( 非モデル生物 ) | 20M~30Mリード |
| Resequencing | 推定ゲノムサイズのx20~x30 |
| メタゲノム解析 ( 16S rRNA ) | 10万リード以上 |
| De novo DNA-seq ( Bacteria ) | 推定ゲノムサイズのx100 |
| De novo DNA-seq ( Long-read ) |
推定ゲノムサイズのx30以上 (ゲノムサイズにより対応不可の場合があります。) |
過去に実施した解析の規模
RNA-seq
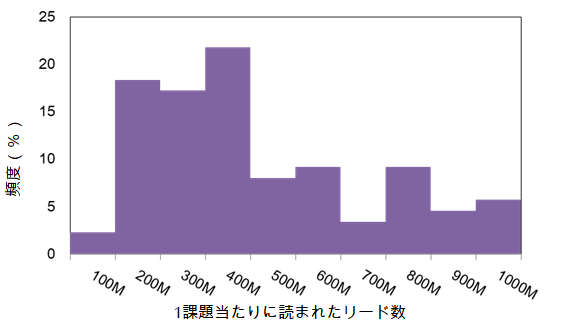
これまでに採択された1課題当たりのリード数の分布です。
ロングリードシーケンス
| 生物種 | N50長 | データ量 | フローセル数 |
| チョウ目の一種 | 11 Kb | 25 Gb | MinION 2枚 |
| インゲン1品種 | 26 Kb | 24 Gb | MinION 1枚 |
| トマト4品種 | 27 Kb~34 Kb | 6 Gb~21 Gb | MinION 各1枚 |
| アブラナ科植物 | 38 Kb | 67 Gb | PromethION 1枚 |
※ サンプルのクオリティに依存します。

